
Hem estudiat i descarregat la proteïna amb Codi PDB 1FOS a la web que podeu veure en la imatge següent:

1FOS és un dímer és a dir, dues proteïnes unides anomenades C-FOS i C-JUN i la seva funció és ser un factor de transcripció que com està format per leucines (aminoàcids) s'uneix en forma d'hèlix a l'ADN i provoca la multiplicació cel·lular i, per tant, el càncer especialment càncer d'ossos i d'endometri.
Aquesta proteïna volem bloquejar-la per impedir el càncer i mesurar l'energia d'unió en kilocalorìes-mol com més negatiu sigui les kilocalories-mol més bo serà moc anticancerigen.
Aquests són els resultats obtinguts de acoplament molecular a l'institut suis de bioinformàtica amb el software Swissdock









Aquí tens les dues gràfiques amb els resultats de la combinació de la proteïna 1FOS amb els diferents compostos, es pot veure que el compost més actiu és el taxol:
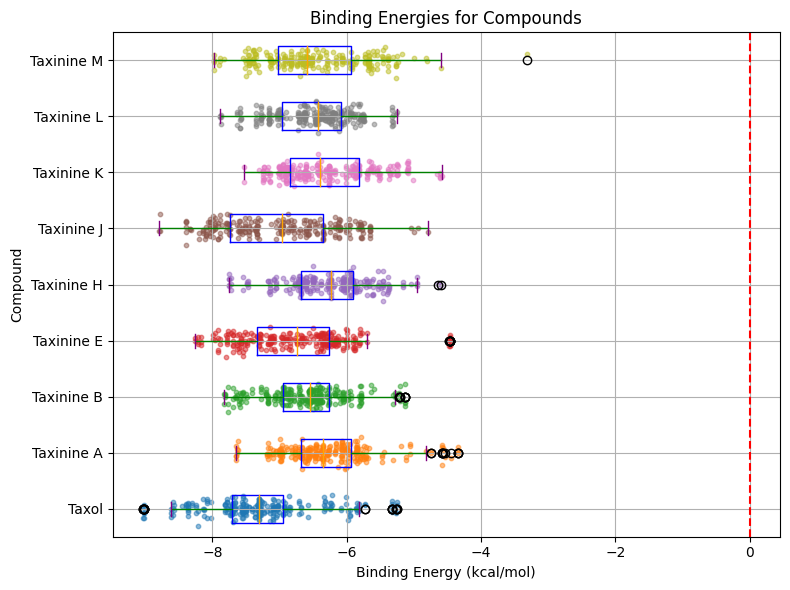

Les conclusions que he obtingut és que el compost més actiu sobre la proteïna 1FOS és el taxol amb una energia d'unió de -9,04 kcal/mol, i això es pot veure en els gràfics.
Abans de posar el codi expliquem que està fet en google.colab on puc triar python o R i CPU O GPU en nootebook settings. La GPU és més útil en deeplearning perquè permet processament paral·lel o simultani de codi i la CPU és la unitat de processament del computador i pot tenir diversos nuclis, però és més lenta en deepearning o visió per ordinador.
Si vull descarregar al google.colab ho puc fer i es descarregarà en format IPYNB que significa interactive python notebook. Aquest arxiu es pot obrir amb un editor Jupyter que es pot instal·lar en ordinador descarregant Anaconda. Hi ha dues versions Jupyter: Jupyter Notebook i Jupyter Lab que es poden descarregar localment en l'ordinador i tenen el mateix aspecte i funcionalitat que google.colab amb l'avantatge i inconvenient d'utilitzar totes les capacitats del teu ordinador.
Aquí tens el codi que ha generat els dos gràfics de més amunt:
# 2. Upload deltaG_values.csv of every docking to generate a boxplot to obtain a summary table transposed data in csv and boxplot
# Has de pujar els diferents arxius i penjar cancel quan acabis, posar els noms en català (en aquest exemple: Taxinina A, etc)
# Després has d'escriure el codi PDB de la teva proteïna (en aquest exemple 1FOS)
# Els signes d'exclamació o "bang=!" indican que s'executen com si estigués f en un terminal o un shell, per exemple: !pwd que significa mostra el directori actual, !cd: change directory, !ls: fes un llistat del derictory on estigui, !ls -l :vol dir que té el paràmetre long és a dir que no només em digui els arxius sinó que també els permisos que poden ser r read(llegibles) w(write) x(executable) de forma que un arxiu dirà: arxiu.csv rwxrw-r-- i això significa el superusuari o administrador té tots els permisos, un usuari no es podrà llegir o escriure i un invitat només podrà llegir. Per canviar permisos la instrucció chmod que significa change mode i vol dir un arxiu que no podem llegir o executar ara es podrà executar. La lletra r té un valor 1, la w té un valor 2 i la x un valor 4 quan escric chmod777 que passa es transforma en rwxrwxrwx és a dir administrador, usuari i invitat tenen els mateixos permisos. I l'última cosa si posem chmod 543 l'administrador té permís 5 --> executar 4 i que pot llegir 1 (4+1=5), l'usuari només pot executar 4, però no pot llegir i l'invitat només pot escriure 2 i llegir 1 (2+1=3), !ls -a: veure tots els arxius perquè a vol dir all, !ls -la: buscar tots els arxius inclosos els ocults i mostrar els seus permisos, !mkdir: Crea make directory amb el nom que posem a continuació nom arxiu i !rm: Remove elimina l'arxiu. Es poden posar !! que el que farien és executar a la cel·la i mostrar la sortida.
!pip install pandas matplotlib io numpy
#Aqui instalem les biblioteques python, pandas ens permet treballar amb csv facilment, matplotlib ens peermet dibuixar grafics, io ens permet entrades (imput) i sortides
from google.colab import files
import pandas as pd
import matplotlib.pyplot as plt
import io
import numpy as np
# Initialize an empty list to store DataFrame objects
dfs = []
# Upload CSV files one by one
print("Upload CSV files one by one. Press Cancel to stop uploading.")
while True:
uploaded_files = files.upload()
if len(uploaded_files) == 0:
break
for filename, contents in uploaded_files.items():
# Read CSV file as DataFrame and append it to the list
df = pd.read_csv(io.StringIO(contents.decode('utf-8')), header=None)
# Add a column to identify the compound
df['Compound'] = f'Compound {chr(ord("A") + len(dfs))}'
dfs.append(df)
# Concatenate DataFrames vertically
combined_df = pd.concat(dfs, ignore_index=True)
# Transpose the DataFrame so that rows become columns
transposed_df = combined_df.set_index('Compound').T
# Save the transposed DataFrame to a new CSV file
transposed_csv_path = 'transposed_data.csv'
transposed_df.to_csv(transposed_csv_path)
# Prompt the user to enter real chemical names for each compound
real_names_mapping = {}
for i, df_name in enumerate(transposed_df.columns):
real_name = input(f"Enter the real chemical name for {df_name}: ")
real_names_mapping[df_name] = real_name
# Prompt the user to enter the last word of the graph title
graph_title_suffix = input("Enter the last word of the graph title: ").strip()
# Create a customized boxplot for compounds
plt.figure(figsize=(8, 6))
# Set colors
box_color = 'blue'
median_color = 'orange'
whisker_color = 'green'
cap_color = 'purple'
# Create a boxplot
boxprops = dict(color=box_color)
medianprops = dict(color=median_color)
whiskerprops = dict(color=whisker_color)
capprops = dict(color=cap_color)
boxplot = transposed_df.boxplot(vert=False, return_type='dict', boxprops=boxprops, medianprops=medianprops, whiskerprops=whiskerprops, capprops=capprops)
# Overlay individual data points
for df_name in transposed_df.columns:
y = np.random.normal(list(transposed_df.columns).index(df_name) + 1, 0.1, size=len(transposed_df[df_name]))
plt.scatter(transposed_df[df_name], y, alpha=0.5, s=10)
# Set ticks and labels
plt.yticks(np.arange(1, len(transposed_df.columns) + 1), [real_names_mapping[col] for col in transposed_df.columns])
plt.xlabel("Energia d'unió (kcal/mol)")
plt.ylabel("Lligands")
plt.title(f"Acoblament molecular amb proteïna PDB {graph_title_suffix}")
plt.grid(True)
plt.axvline(x=0, color='red', linestyle='--') # Add line at 0 for reference
plt.tight_layout()
# Save the plot as an image file
plot_image_path = 'boxplot.png'
plt.savefig(plot_image_path)
# Download the transposed CSV file and the plot image
files.download(transposed_csv_path)
files.download(plot_image_path)
# Print paths to the saved files
print("Transposed data saved to:", transposed_csv_path)
print("Plot image saved to:", plot_image_path)